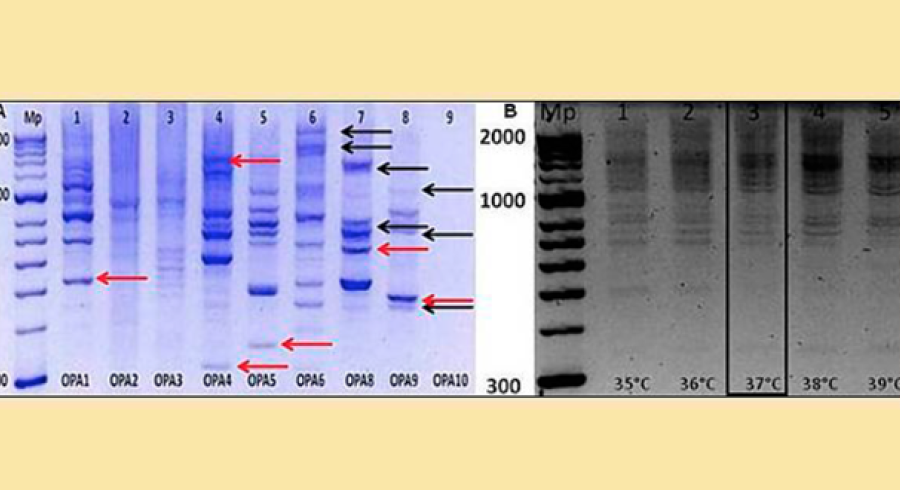
Se implementó el método Taguchi para optimizar marcadores RAPD-PCR que permitan estimar la diversidad genética con el modelo: tortuga cabezona. Se aisló ADN de C. caretta de dos zonas del Caribe colombiano (Don Diego N = 5 e Islas del Rosario N = 3) y se cuantificó. Se aplicó una matriz ortogonal de Taguchi para estandarizar cuatro variables para la reacción RAPD-PCR. Los datos obtenidos se analizaron con el programa PopGen. Las condiciones estandarizadas se encontraron con 7,85 ng/μl de ADN, 3,5 mM de MgCl2, 200 mM de dNTP´s, 0,5 μM de oligonucleótido y una unidad de Taq ADN polimerasa, en un volumen final de reacción de 20 μl. Las condiciones de termociclado iniciaron a 94°C por 5 m, seguido de 40 ciclos de: 94°C por 40 s, 37°C por 40 s y 72°C por 90 s. Los marcadores se registraron en una matriz binaria de presencia (1) y ausencia (0), y como un ejemplo modelo se determinó la diversidad genética utilizando el índice de Shannon (H´= 0,44+/–0,27 individuos de Don Diego y de H´= 0,25+/–0,32 para Isla del Rosario), el índice promedio de estructura genética (Gst = 0,27) y el índice de migración efectiva (Nm = 1,28). Se estandarizó una metodología haciendo uso del método de Taguchi que produce bandas claras, legibles y reproducibles, método que puede emplearse como alternativa confiable para realizar estudios de diversidad genética en la tortuga cabezona o de otras especies, y adicionalmente, integrarlos en el currículo de biología molecular y/o bioquímica para estudiantes de pregrado y maestría.
Método Taguchi para optimizar marcadores RAPD-PCR y determinar diversidad genética: un modelo, la tortuga cabezona Caretta caretta (Testudines: Cheloniidae)
Artículo de la Revista Mutis Vol.3, N°1
Artículo
Gratuita
Formato:
Impreso